bioslawek
Dołączył: 09 Sty 2015
Posty: 96
Przeczytał: 0 tematów
Płeć: Mężczyzna
|
 Wysłany: Sob 12:47, 21 Sie 2021 Temat postu: Pseudogeny: genetyczne śmieci czy sekwencje funkcjonalne? Wysłany: Sob 12:47, 21 Sie 2021 Temat postu: Pseudogeny: genetyczne śmieci czy sekwencje funkcjonalne? |
|
|
Pseudogeny: Pseudo-funkcjonalne czy kluczowe regulatory w zdrowiu i chorobie?
„Pseudogeny od dawna są określane jako „śmieciowe” DNA, nieudane kopie genów, które powstają podczas ewolucji genomów [lub takie, które po awarii wyłączają całe sieci genetyczne – np. te, które rzekomo przyczyniły się do utraty zębów u ptaków czy mrówkojadów].

Jednak ostatnie wyniki badań podważają ten pseudogenizm; w rzeczywistości niektóre pseudogeny wydają się mieć potencjał do regulowania swoich kodujących białka kuzynów [genów]. Dalekie są od bycia cichymi reliktami, wiele pseudogenów ulega transkrypcji do mRNA, niektóre wykazują specyficzny wzór aktywacji w tkankach.
Transkrypty pseudogenów mogą być porzetwarzane w krótkie, interferujące RNA, które regulują ekspresję kodujących genów, poprzez szlak RNAi.
W innym niezwykłym odkryciu wykazano, że pseudogeny są zdolne do regulowania supresorów nowotworowych i onkogenów, działając jako wabiki mikroRNA. Odkrycie, że pseudogeny często ulegają deregulacji podczas progresji nowotworu, gwarantuje dalsze badanie prawdziwego zakresu funkcji pseudogenów.
Genom ludzki, podobnie jak u innych ssaków, jest zaśmiecony różnorodnymi powtarzającymi się elementami i niekodującymi genami. Jednym z takich elementów jest pseudogen, zaburzone mutacjami punktowymi i pocięte delecjami odwzorowanie oryginalnego genu kodującego białko, który utracił zdolność do wytwarzania funkcjonalnego białka. Ponieważ nie kodują białek, często zakłada się, że pseudogeny są niefunkcjonalne i zaliczane do „śmieciowego DNA”.
Podczas gdy niektóre pseudogeny są ciche transkrypcyjnie, inne są aktywne, co rodzi pytanie, czy ich niekodujące transkrypty bezsensownie zużywają energię komórki, czy zamiast tego są wykorzystywane przez komórkę do regulacji kodujących genów. To pytanie jest szczególnie istotne, biorąc pod uwagę niedawną lawinę dowodów sugerujących, że długie niekodujące mRNA odgrywają kluczową rolę w regulacji funkcji genomu.Pseudogeny mogą powstawać poprzez różne mechanizmy. Spontaniczne mutacje w genie kodującym, które uniemożliwiają transkrypcję lub translację genu – prowadzą do powstania „jednolitego” pseudogenu . Zduplikowane pseudogeny są tworzone przez duplikację tandemową, lub nierówny crossing-over. Te zduplikowane geny tracą swój potencjał kodowania białek z powodu utraty promotorów, lub wzmacniaczy, albo okaleczających je mutacji, takich jak przesunięcia ramki odczytu, lub powstawanie przedwczesnych kodonów stop.
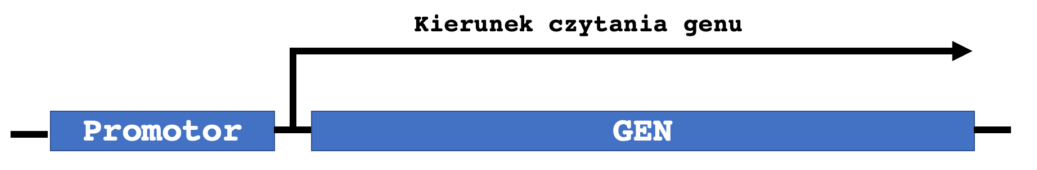
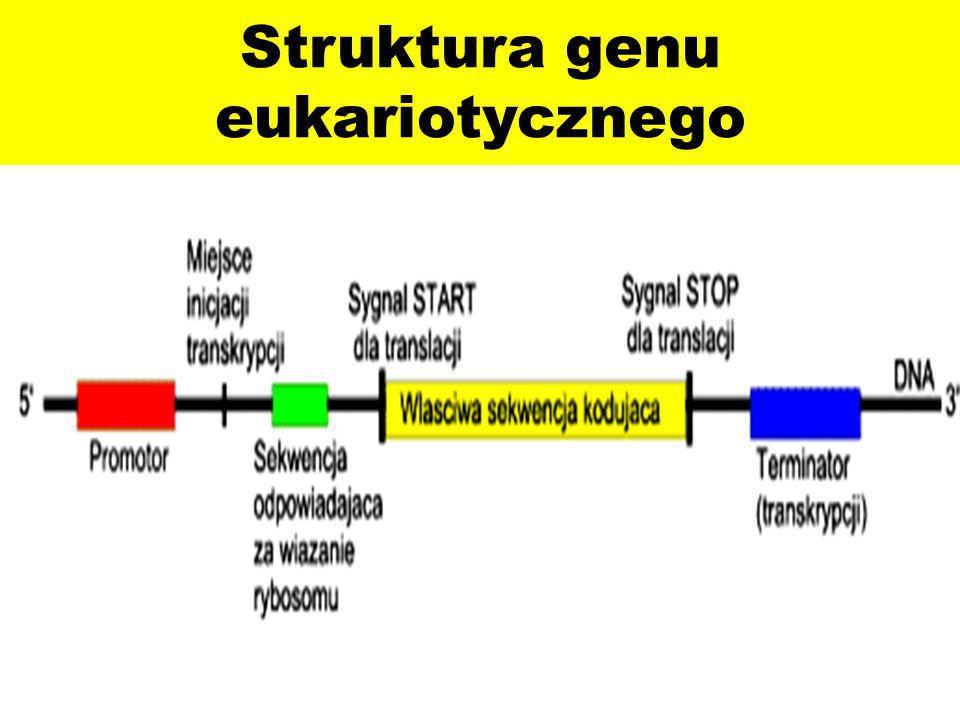
Jednak mają tendencję do zachowywania swojej charakterystycznej struktury intron-egzon. W przeciwieństwie do tego, retrotransponowane, lub „przetworzone” pseudogeny (PP) są wytwarzane, gdy transkrypt mRNA jest poddawany odwrotnej transkrypcji i integrowany z genomem w nowej lokalizacji, a zatem w tym przypadku nie zawierają intronów.Inne wspólne cechy PP to ich ciągi poliA i bezpośrednie powtórzenia na każdym końcu pseudogenu. Wydaje się, że w retrotranspozycji mRNA do DNA i integracji z genomem pośredniczy długi, rozproszony element jądrowy 1 (L1), a aktywność transkrypcyjna powstałego PP zależy od tego, czy zdarzenie integracji zachodzi blisko innego promotora. Zbiór przetworzonych pseudogenów w ludzkim genomie został wygenerowany z zaledwie 10% genów kodujących. Wysoce eksprymowane geny porządkowe z większym prawdopodobieństwem produkują PP, podobnie jak inne krótsze RNA o wysokim stopniu transkrypcji. Przykładem jest niewielka liczba genów kodujących białka rybosomalne, które stanowią około 20% ludzkich PP .Termin pseudogen został ukuty w 1977, kiedy Jacq i współpracownicy odkryli wersję genu kodującego 5S rRNA, który został skrócony, ale zachował homologię z aktywnym genem Xenopus laevis. W ciągu następnych dwóch dekad pseudogeny odkryto sporadycznie. Przyspieszenie technologii sekwencjonowania nowej generacji w połączeniu z projektem senkwencjonowania ludzkiego genomu pozwoliło uzyskać obrazy pełnego genomu szeregu organizmów, umożliwiając znacznie dokładniejsze analizy częstości występowania pseudogenów.
Co godne uwagi, pseudogeny są prawie tak liczne jak geny kodujące, a prognozy wahają się od 10 000 do 20 000 ludzkich pseudogenów. Większość ludzkich pseudogenów to PP, podczas gdy liczba jednostkowych pseudogenów w ludzkim genomie wynosi <100.Pseudogeny są obecne w wielu gatunkach, w tym roślinach, bakteriach – choć nie są tak liczne w organizmach jednokomórkowych w owadach i nicieni , ale są one szczególnie liczne u ssaków.Pseudogeny są czasami uważane za reprezentujące „sekwencję neutralną”, w której kumulujące się mutacje nie są selekcjonowane szkodząc, lub wspierając organizm – skoro miały nie mieć wpływu na fenotyp, to tym samym z punktu widzenia darwinizmu dobór naturalny ich nie zauważa. Jednak ta przesłanka opiera się na założeniu, że pseudogeny są funkcjonalnie obojętne. Ostatnio pojawiły się dowody na to, że niektóre pseudogeny są funkcjonalnie aktywne, a zatem badanie ich ewolucji i przyczyn konserwowania przez naturalną selekcję może wesprzeć przekonanie o ich użyteczności dla organizmu dać wgląd w ich potencjalny mechanizm działania.
[….]
Przecinek, Stare czasy panie January, ale już pan ma wyjaśnienie i to nie ze strony kreacjonistów! 🙂
[link widoczny dla zalogowanych]
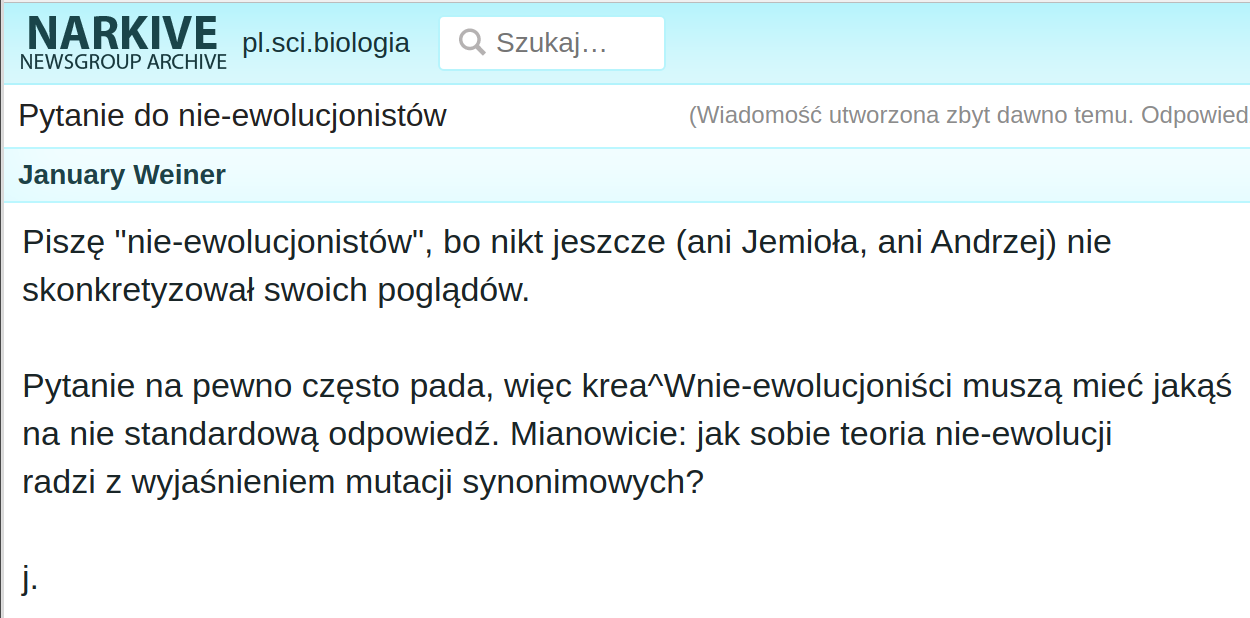
Mutacja synonimiczna
Mutacja synonimiczna to zmiana pojedynczego nukleotydu w genie (mutacja punktowa) nie powodująca zmiany aminokwasu w kodowanym białku ze względu na to, że kod genetyczny jest zdegenerowany, czyli jeden aminokwas może być kodowany przez kilka kodonów (np. zmiana CCC na CCU nie powoduje zmiany aminokwasu, gdyż obie trójki kodują prolinę). Wydaje się, że w niektórych przypadkach, jeśli zmiana następuje z kodonu preferowanego na rzadki, może to wpłynąć na szybkość syntezy białka, a to z kolei na szybkość jego zwijania, a zatem na jego strukturę przestrzenną, co może mieć wpływ na fenotyp
[link widoczny dla zalogowanych]

[….]
Niedawno doniesiono, że synonimowe mutacje punktowe występują znacznie częściej niż niesynonimowe zmiany zasad w pseudogenie Drosophila Est-6 .
U kur zaobserwowano, że w obrębie wielu IglV i IghVpseudogenów, liczba kodonów stop zawartych w sekwencji „kodującej” jest znacznie niższa niż można by się spodziewać, gdyby podstawienia nukleotydów występowały losowo; ponadto większość kodonów stop wprowadzonych przez mutacje punktowe jest następnie „korygowana” i eliminowana przez kolejne mutacje punktowe w tym samym kodonie.
Ta cecha, która występuje również w V H pseudogenów u myszy, może wskazywać, że te pseudogeny są w stanie kodować białka, lub że ochrona otwartych ramek odczytu umożliwia pseudogenom zaangażowanie w rearanżacje genów somatycznych.
Pseudogeny stopniowo gromadzą mutacje, a liczba mutacji może dać nam oszacowanie ich wieku. (….) Analiza regionu rozszerzonego układu zgodności tkankowej makaka (MHC) klasy II ujawniła dwa pseudogeny, które okazały się homologiczne do ludzkich pseudogenów HIV typu TAT-podobnych do czynnika 1 i palca cynkowego, co sugerowało konserwację ewolucyjną tych sekwencji.
Badania prowadzone przez Podlaha i współpracowników wykazały, że pseudogen Makorin1-p1 jest zachowany w szczepach Mus musculus i Mus pahari . To skłoniło specjalistów do badania całych genomów i poszukiwania pseudogenów zachowanych między ludźmi a myszami. Ludzkie pseudogeny, wraz z ich genami macierzystymi, zostały porównane z odpowiadającymi im ortologami myszy i ich pseudogenami.
Co ciekawe, stwierdzono, że wiele z badanych pseudogenów nagromadziło bardzo niewiele mutacji w regionach regulatorowych w porównaniu z genami macierzystymi, co może sugerować, że te regiony regulatorowe mają znaczenie dla pseudogenu i że pseudogen może być funkcjonalny.
Spośród badanych grup genów i pseudogenów analiza sekwencji sugerowała, że 30 z nich reprezentuje pseudogeny, które były obecne zarówno u myszy, jak i u ludzi i powstały przed rozdzieleniem się tych dwóch gatunków. Porównanie transkrybowanych ludzkich pseudogenów pokazuje, że 50% jest konserwatywnych u rezusów, ale tylko 3% jest konserwatywnych u myszy.
Analizy tych pseudogenów wykazały, że pomimo K A / K S szybkość substytucji wskazująca na niekodujące RNA, poziomy GC i szybkość mutacji w tych pseudogenach jest ograniczona w stosunku do otaczających je regionów międzygenowych. Są to liczne dowody na to, że niektóre pseudogeny wykazują funkcjonalną rolę w organizmach, które je przechowują.
Większość pseudogenów traci zdolność do transkrypcji, albo z powodu mutacji w ich promotorze, albo (w przypadku PP) integracji z cichymi regionami genomu. Dokonywanie dokładnych pomiarów transkrypcji pseudogenów jest skomplikowane ze względu na podobieństwo, jakie dzielą one z genami macierzystymi .
Jednakże istnieje wiele przykładów pseudogenów, które podlegają transkrypcji, w tym pseudogeny dla supresora nowotworu PTEN (którego transkrypty są liczniejsze niż gen macierzysty), nadnerczowej hydroksylazy sterydowej P450c21A, ludzki interferon leukocytów, GAPDH , glukocerebrozydaza i Oct4. Technologia mikromacierzy umożliwia analizę transkrypcji pseudogenów na znacznie większą skalę. Szacunki dotyczące proporcji transkrybowanych ludzkich pseudogenów wahają się od 2% do 20%.
Badanie wzorca transkrypcji w tkankach i liniach komórkowych może dać wgląd w potencjalną funkcjonalność. Inne niekodujące RNA wykazują specyficzne dla tkanki wzorce ekspresji i wykazano również, że pełnią role funkcjonalne, w tym antysensowne RNA , transkrypty międzygenowe i długie niekodujące RNA oraz miRNA .
W badaniu transkrypcji w regionach ENCODE genomu stwierdzono transkrypcję 14 pseudogenów. Pięć z nich zostało przepisanych wyłącznie w jądrach, a kolejne cztery były również aktywne w jądrach i innych tkankach. Ten wzór transkrypcji jest zgodny z wcześniejszymi wynikami, co sugeruje możliwe znaczenie biologiczne dla specyficznej dla jąder transkrypcji pseudogenów. Istnieje również kilka przykładów pseudogenów, których czasowo-przestrzenny wzorzec ekspresji różni się od wzorca ich genu macierzystego.
Regulacja ekspresji genu - ppt pobierz
Specyficzne zmiany w ekspresji pseudogenów mogą również wystąpić w różnych warunkach fizjologicznych, w tym w chorobach takich jak cukrzyca i rak. Przykłady dynamicznej transkrypcji pseudogenów zaobserwowano w innych organizmach. Silną transkrypcję pseudogenów zmierzono w Mycobacterium leprae, organizmie wywołującym trąd, przy poziomach specyficznych transkryptów zmieniających się podczas procesu infekcji. Indukcja stresu u Arabidopsis thaliana prowadzi do zmian w ekspresji wielu genów i pseudogenów.
Aktywność transkrypcyjna pseudogenu będzie częściowo zależeć od wykorzystywanego przez niego promotora. Niektóre mają własne promotory, podczas gdy inne wykorzystują promotory pobliskich genów. Przetworzony pseudogen jest w dużej mierze na łasce swojego miejsca integracji, jeśli chodzi o aktywność promotora. Dlatego też różnica we wzorcu transkrypcji między pseudogenem a jego genem macierzystym niekoniecznie odzwierciedla rolę funkcjonalną, ale zamiast tego może być jedynie wynikiem kierowania przez nowy promotor.
Gdyby to ostatnie było prawdą, można by przewidzieć, że akt transkrypcji pseudogenu byłby ewolucyjnie neutralny (lub nawet wybrany przeciwko oszczędzaniu energii komórkowej). Analiza konserwatywnych pseudogenów transkrybowanych pokazuje, że około 50% jest rzeczywiście zachowanych przez miliony lat ewolucji naczelnych (chociaż znacznie mniej jest zachowanych między gatunkami bardziej odległymi od człowieka, takimi jak gryzonie).
Fakt, że transkrypcja niektórych pseudogenów jest specyficzna tkankowo, dynamiczna i utrzymywana od tysiącleci sugeruje, że ich transkrypty mogą odgrywać pewną funkcjonalną rolę w komórkach. Nic nie zastąpi jednak eksperymentów funkcjonalnych, aby sprawdzić, czy pseudogeny i ich transkrypty mają bezpośrednią aktywną rolę.
Wydaje się, że wiele organizmów wielokomórkowych zachowuje obecność pseudogenów i nakłada ograniczenia na same sekwencje – uniemożliwia ich ekspresję. Z drugiej strony, różne organizmy jednokomórkowe aktywnie wydalają geny, które uległy pseudogenizacji. Pozostaje zatem pytanie, jaka jest potencjalna korzyść dla ssaków i innych złożonych organizmów z zatrzymywania i prawdopodobnie obejmowania ochroną genów, które utraciły potencjał kodowania białek?
Jedną z proponowanych funkcji pseudogenów jest działanie jako źródło różnorodności genetycznej, na przykład w wytwarzaniu przeciwciał lub zmienności antygenu. Jednak kontekście niekodującego RNA pseudogeny oferują potencjalnie znacznie bardziej dynamiczne mechanizmy regulacji dynamicznych procesów jądrowych. W ostatniej dekadzie ujawniono nowy poziom złożoności w regulacji ekspresji genów i funkcji jądra. Wykazano, że wiele niekodujących RNA, zarówno długich, jak i krótkich, reguluje różne procesy w komórkach. Wydaje się, że niekodujące RNA wytwarzane z niektórych pseudogenów są wykorzystywane przez różne fascynujące mechanizmy do kontrolowania funkcji genów.
(….)
W dwóch przełomowych doniesieniach wykazano, że w mysich oocytach części wielu transkryptów pseudogenów są przetwarzane na małe interferujące RNA (siRNA). Te siRNA pochodziły z pseudogenów o wewnętrznej strukturze drugorzędowej lub z dsRNA składających się z sensownych i antysensownych transkryptów (zaobserwowano kombinacje mRNA pseudogen-pseudogen i kodujący pseudogen). Utrata Dicer (białka niezbędnego do produkcji siRNA) doprowadziła do spadku poziomów siRNA pochodzących z pseudogenów i wzrostu poziomu kodowania mRNA genów z homologią z sekwencjami siRNA.
Sugeruje to, że siRNA wytworzone z dsRNA są zdolne do tłumienia ekspresji genów. Na przykład siRNA wygenerowane ze struktury spinki do włosów w pseudogenowym RNA Au76 były w stanie zahamować ekspresję z rodzicielskiego genu kodującego Rangap1 ( Watanabe i wsp. 2008). Wykryto kilka siRNA niosących podobieństwo sekwencji do kompleksu deacetylazy histonowej, HDAC1.
Wszystkie sekwencje siRNA pochodziły z serii pseudogenów HDAC1 (brak z samego genu HDAC1 ), jednak po nokaucie Dicer poziomy mRNA HDAC1 wzrosły, co sugeruje, że gen kodujący jest regulowany przez kompleks wyciszający indukowany przez RNA (RISC). ). W innym przypadku siRNA wytworzono z regionów dsRNA utworzonych między mRNA Ppp4r1 a antysensownym RNA wytworzonym z pseudogenu o 90% homologii.
siRNA wytworzone z tego parowania wydają się tłumić Ppp4r1gen. Podobne badanie na gatunku ryżu pokazuje, że niewielka część pseudogenów ulega transkrypcji i przetwarzaniu w siRNA po sparowaniu z genem kodującym lub paralogicznym transkryptem pseudogenu. Odkrycia te sugerują potencjalny mechanizm działania transkryptów antysensownych. Jednak okaże się, czy podobne procesy zachodzą w komórkach somatycznych ssaków.
(…..)
miRNA to klasa niekodujących RNA, które wpływają na stabilność mRNA. W ich specyficzności i funkcji pośredniczy parowanie zasad z celem (głównie w 3′ UTR); ich głównym efektem jest powodowanie degradacji mRNA, a tym samym obniżanie poziomów ekspresji. W niedawnym raporcie wykazano, że para gen-pseudogen jest koregulowana przez te same miRNA.
PTEN jest supresorem guza, który często ulega mutacji w jednym allelu w momencie pojawienia się raka. Podatność na raka zależy od dawki PTEN. Dlatego utrzymanie precyzyjnych poziomów białka PTEN ma kluczowe znaczenie dla zapobiegania onkogenezie. PTENP1 , pseudogen supresora nowotworu PTEN , podlega transkrypcji na wysokim poziomie.
(……)
Sugeruje to, że pseudogen PTENP1 działa jako „wabik miRNA”, wiążąc się i tym samym zmniejszając efektywne stężenie komórkowe miRNA, umożliwiając tym samym ucieczkę PTEN z represji, w której pośredniczy miRNA. Funkcjonalne powiązanie między parą PTEN / PTENP1 jest zgodne z odkryciami, że ich poziomy są często skorelowane w próbkach raka prostaty i że delecje ogniskowe zawierające PTENP1 występują często w sporadycznych przypadkach raka okrężnicy.
Podobny związek wykazano również między onkogenem KRAS a jego pseudogenemKRASP1. Co ciekawe, zdolność pseudogenu HMGA1-p do destabilizacji mRNA HMGA1 zależy od regionu 3′ UTR, co sugeruje, że zarówno pozytywne, jak i negatywne czynniki stabilizacji są zdolne do konkurencyjnej interakcji z nieulegającymi translacji regionami genów i pseudogenów do regulowania wydajności ekspresji.
(…..)
W dużej mierze pseudogeny zostały przeoczone w dążeniu do zrozumienia biologii zdrowia i choroby do tego stopnia, że sondy pseudogenów są często nieobecne w dostępnych na rynku mikromacierzach. Ponieważ pojawiają się dowody, że pseudogeny ulegają deregulacji w chorobie i że ich deregulacja może przyczyniać się do chorób takich jak cukrzyca i rak, powszechne nastawienie, że są to niefunkcjonalne relikty, powoli się zmienia.
Wraz z pojawieniem się niedrogiego sekwencjonowania nowej generacji, badania transkryptomiki, a w szczególności pseudogenów (i innych transkrybowanych elementów niekodujących), powinny doświadczyć kwantowego skoku naprzód. W nadchodzącym dziesięcioleciu zakres i mechanizmy funkcji pseudogenów powinny stać się jaśniejsze.”
ŹRÓDŁA
[link widoczny dla zalogowanych]
Regulacja ekspresji genu
[link widoczny dla zalogowanych]
[link widoczny dla zalogowanych]
Tajemnicze 98% ludzkiego genomu: naukowcy chcą rzucić światło na tą część ludzkiego genomu, która jest przyrównywana do ciemnej materii
Po zakończeniu w 2003 roku Projektu Ludzkiego Genomu – który zsekwencjonował wszystkie 3 miliardy „ liter ” lub par zasad w ludzkim genomie – wielu myślało, że nasze DNA stanie się otwartą księgą. Szybko jednak pojawił się kłopotliwy problem: chociaż naukowcy mogli dokonać transkrypcji książki, mogli zinterpretować tylko niewielki jej procent.
Tajemnicza większość – aż 98 procent – naszego DNA nie koduje białek. Uważa się, że większość tego „genomu ciemnej materii” to niefunkcjonalne resztki ewolucyjne, które dopiero czekają na przejażdżkę. Jednak wśród tego niekodującego DNA kryje się wiele kluczowych elementów regulacyjnych, które kontrolują aktywność tysięcy genów. Co więcej, pierwiastki te odgrywają ważną rolę w chorobach, takich jak rak, choroby serca i autyzm, i mogą stanowić klucz do możliwych wyleczeń.
Dowód na jednorazową utratę zmineralizowanych zębów u wspólnego przodka ptaków
[link widoczny dla zalogowanych]
Struktura i funkcja ameloblastyny jako białka macierzy zewnątrzkomórkowej: adhezja, wiązanie wapnia i interakcja z CD63 u ludzi i myszy
Ludzie i ptaki mają te same geny odpowiedzialne za uczenie się mowy i śpiewu
[link widoczny dla zalogowanych]
Analiza genomowa ujawnia plejotropowe allele w EDN3 i BMP7 zaangażowane w kolor grzebienia kurczaka i produkcję jaj
Identyczne krótkie sekwencje peptydowe w niepowiązanych białkach mogą mieć różne konformacje: poligon testowy dla teorii rozpoznawania immunologicznego
Ewolucja zbieżna w elementach strukturalnych białek badanych za pomocą analizy profili krzyżowych
[link widoczny dla zalogowanych]
Białka o tej samej fałdzie i niepowiązanych sekwencjach mają podobny skład aminokwasowy
[link widoczny dla zalogowanych]
Głęboka homologia w erze sekwencjonowania nowej generacji
[link widoczny dla zalogowanych]
Jak różne sekwencje aminokwasów determinują podobne struktury białek: Struktura i dynamika ewolucyjna globin
[link widoczny dla zalogowanych]
Wykrywanie adaptacyjnej, zbieżnej ewolucji aminokwasów
[link widoczny dla zalogowanych]
W genomice ewolucyjnej naukowcy zainteresowali się identyfikacją substytucji, które przyczyniają się do zbieżnych adaptacji fenotypowych. Jest to trudne pytanie, które wymaga odróżnienia zbieżnych podstawień pierwszego planu, które są zaangażowane w zbieżny fenotyp od tła zbieżnego podstawienia. Mogą być one powiązane z innymi adaptacjami, mogą być neutralne lub mogą być konsekwencją błędów mutacyjnych. Ponadto nie ma ogólnie przyjętej definicji zbieżnych podstawień. W literaturze zaproponowano różne metody, które wykorzystują różne definicje, co skutkuje różnymi zestawami kandydujących zbieżnych podstawień pierwszego planu. W tym artykule najpierw opisujemy procesy, które mogą generować zbieżne podstawienia pierwszego planu w sekwencjach kodujących, oddzielając procesy adaptacyjne od nieadaptacyjnych. Po drugie, dokonujemy przeglądu metod, które zostały zaproponowane do wykrywania zbieżnych podstawień pierwszego planu w sekwencjach kodujących i ujawniają założenia, które leżą u ich podstaw.
FOXP2
[link widoczny dla zalogowanych]
Koncepcja ewolucyjna w genetyce i genomice
[link widoczny dla zalogowanych]
[link widoczny dla zalogowanych]
Istnieje wiele przekonujących przykładów zbieżności molekularnej poszczególnych genów. Jednak częstość występowania i względne znaczenie adaptacyjnej konwergencji całego genomu pozostają w dużej mierze nieznane. Wiele ostatnich prac donosiło o uderzających przykładach nadmiernej konwergencji całego genomu, ale niektóre z tych badań zostały zakwestionowane z powodu użycia niewłaściwych modeli zerowych. Tutaj zsekwencjonowaliśmy i porównaliśmy genomy 12 gatunków jaszczurek, które niezależnie zbiegły się w zestawach adaptacyjnych cech behawioralnych i morfologicznych. Pomimo szeroko zakrojonych poszukiwań sygnatury konwergencji molekularnej obejmującej cały genom, nie znaleźliśmy dowodów potwierdzających konwergencję molekularną określonych aminokwasów ani w poszczególnych genach, ani w porównaniach w całym genomie; nie odkryliśmy również żadnych dowodów potwierdzających nadmiar adaptacyjnej konwergencji w szybkościach podstawień aminokwasów w genach. Nasze odkrycia wskazują, że kompleksowa konwergencja fenotypowa nie znajduje odzwierciedlenia na poziomie kodowania białek u anoli w całym genomie, a zatem ta adaptacyjna konwergencja fenotypowa prawdopodobnie nie jest ograniczana przez ewolucję wielu specyficznych sekwencji białkowych lub struktur.
[link widoczny dla zalogowanych]
”Ewolucja zbieżna odnosi się do ewolucji w różnych rodach struktur, które są podobne lub „analogiczne”, ale nie można tego przypisać istnieniu wspólnego przodka; innymi słowy, fakt, że struktury są analogiczne, nie odzwierciedla homologii. Podobieństwo może leżeć na poziomie fenotypowym, w którym to przypadku linie mają tę samą jawną cechę, ale podstawowe sekwencje DNA są różne.
Ewolucja zbieżna zachodzi, gdy gatunki zajmują podobne nisze ekologiczne i adaptują się w podobny sposób w odpowiedzi na podobne presje selekcyjne. Cechy, które powstają w wyniku zbieżnej ewolucjisą określane jako „struktury analogiczne”. Kontrastowane są ze „strukturami homologicznymi”, które mają wspólne pochodzenie. Przeciwieństwem ewolucji konwergentnej jest ewolucja rozbieżna, w której spokrewnione gatunki rozwijają różne cechy.
Dobrze udokumentowane przypadki zbieżnej ewolucji podobnych sekwencji DNA nie są liczne; takie przypadki są zwykle ograniczone do kilku aminokwasów. Ewolucja zbieżna może wprowadzać w błąd biologów zajmujących się filogenezą, ponieważ naśladuje wspólne pochodzenie. Standardowe metody filogenetyczne nie są przystosowane do rozróżnienia między nimi. Kiedy ewolucja zbieżna jest mylona z homologią, powstaje drzewo filogenetyczne, które ma zafałszowany wygląd, to znaczy gatunki wydają się pochodzić od wspólnego przodka, podczas gdy w rzeczywistości tak nie jest.”
[link widoczny dla zalogowanych]
[link widoczny dla zalogowanych]
„Chociaż wyniki ENCODE sugerują, że co najmniej 80% „śmieciowego” DNA faktycznie odgrywa rolę w kontrolowaniu innych genów, Birney uważa, że bardziej prawdopodobne jest, że prawie 100% z niego okaże się mieć cel, a wiele innych genów regulacyjnych czeka na odkrycie. ”
[link widoczny dla zalogowanych]
Pilnie potrzebne są zmiany paradygmatu w poszukiwaniu nowych leków, ponieważ tradycyjna ścieżka odkrywania leków prawie się wyczerpała. Być może w przyszłości sektor farmaceutyczny wykorzysta unikalne mocne strony kilku strategii do opracowania rozwiązań kombinatorycznych. Tylko czas pokaże, czy rozwiązania terapeutyczne oparte na „śmieciowym DNA” przetrwają próby kliniczne. Pomysł wydaje się jednak nowatorski i bardzo interesujący, by zasługiwał na bliższe przyjrzenie się.
Zakres funkcjonalności w ludzkim genomie
John S Matticki
Marcel E Dinger
[link widoczny dla zalogowanych]
[link widoczny dla zalogowanych]
[link widoczny dla zalogowanych]
Nowy artykuł w „Nature Reviews Genetics”, Overcoming Challenges and Dogmas to Understand the Functions of Pseudogenes1 [Pokonywanie wyzwań i dogmatów w celu zrozumienia funkcji pseudogenów], jest po prostu niesamowity. Dokumentuje nie tylko, że pseudogeny pełnią ważne funkcje, ale także że ze względu na obecne „dogmaty” w biologii, jak również na współczesne ograniczenia techniczne ‒ nie rozpoznajemy ich prawdziwych funkcji. Jak ujęli to Seth Cheetham i współautorzy, biologia ucierpiała w wyniku „braku motywacji do badania funkcji pseudogenów z powodu przyjęcia założenia, że nie pełnią one żadnych funkcji”2, przy czym „dominujące ograniczenie w postępach badań pseudogenów ma związek z przekonaniem, że regiony pseudogenów są z natury niefunkcjonalne”3.
W streszczeniu artykułu autorzy bardzo jasno przedstawili swoją tezę:
Pseudogeny są zdefiniowane jako regiony genomu zawierające wadliwe kopie genów. Znajdujemy je w prawie wszystkich formach życia, a w genomach ssaków występują w podobnej liczbie, co rozpoznane geny kodujące białka. Chociaż często przypuszcza się, że nie pełnią one żadnych funkcji, okazuje się, że coraz więcej pseudogenów odgrywa ważne role biologiczne. Biorąc pod uwagę ich ewolucyjne pochodzenie i ograniczenia metod sekwencjonowania genomu, twierdzimy, że pseudogeny zostały błędnie sklasyfikowane. Naszym zdaniem powszechne nieporozumienie dotyczące pseudogenów, utrwalone częściowo przez przypisanie im pejoratywnego określenia „pseudogen”, doprowadziło do częstego wykluczania ich z badań funkcjonalności i z analiz genomowych. W naszej opinii wraz z pojawieniem się technik upraszczających badanie pseudogenów obiektywne spojrzenie na te elementy genomowe umożliwi ujawnienie cennych informacji na temat funkcji i ewolucji genomu4.
Zwracają oni uwagę, że wiele obszarów DNA, które pierwotnie uznano za pseudogenowe śmieci, odgrywa jednak określoną rolę: „zważywszy na to, że rośnie liczba regionów pierwotnie zidentyfikowanych jako pseudogenowe, które później okazały się pełnić funkcje biologiczne, istnieje ryzyko, że pewne regiony genomu są przedwcześnie odrzucane jako pseudogeny i uważane za pozbawione funkcji”5.
Szukajmy funkcji, a je znajdziemy [….]
Rozbieżność ewolucyjna i Konwergencja w białkach
Kod genetyczny
[link widoczny dla zalogowanych]
Struktura białka
[link widoczny dla zalogowanych]
Genetyka oglna wykad dla studentw II roku biotechnologii
[link widoczny dla zalogowanych]
|
|







